Autores: Antonio Olmos (1), Ana Belén Ruiz-García (1), Natàlia Puig (2), Eva Pérez (2), Mireia Bordas (2)
- Laboratorio de Virología de Leñosas del IVIA
- Agromillora Iberia, SLU
España es uno de los principales productores de vino en el mundo, y se trata de un negocio ligado a una gran tradición histórica.
El sector vitivinícola español genera un gran valor económico; con 1.123.644 hectáreas de viñedo -según datos de la FAO en 2018-, España sigue a la cabeza de las superficies cultivadas.
Además, la producción de uva para vinificación en España tiene una gran importancia social, con un volumen de trabajo que va en aumento en los últimos años y que constituye una base para el desarrollo rural. Los viñedos desempeñan también un papel importante en la conservación medioambiental, impidiendo la erosión del suelo. Ambos factores hacen de este cultivo un pilar para la sostenibilidad de nuestra agricultura.
‘Alternativas para una viticultura avanzada y sostenible’
Motivados por ofrecer plantas de vivero de vid de excelente calidad sanitaria, en 2018 Agromillora inició el proyecto CDTI IDI-20180247 ‘Alternativas para una viticultura avanzada y sostenible’. Con más de 25 años de dedicación al sector, esta empresa viverística es pionera en la comercialización de planta injertada de viña procedente del vivero VCR. En el marco de este proyecto se ha establecido una colaboración con el Laboratorio de Virología de Leñosas del Instituto Valenciano de Investigaciones Agrarias (IVIA) para la detección de virus en plantas de vid.
Tradicionalmente, el diagnóstico de infección viral se ha basado en el bioensayo o la técnica serológica ELISA. Sin embargo, ambas técnicas presentan claros inconvenientes relacionados con el espacio/tiempo requerido, la incapacidad de identificar el patógeno (bioensayos) o la ausencia de anticuerpos contra patógenos importantes. La técnica ELISA ha sido uno de los métodos de diagnóstico más comunes, sin embargo puede no detectar infecciones si el virus se encuentra en bajas concentraciones, dando lugar a falsos negativos. Esta técnica también puede originar falsos positivos en el caso de que los anticuerpos policlonales reaccionen inespecíficamente con material vegetal o con otras especies virales.
PCR
En los últimos años, con el avance del conocimiento del genoma de los virus, se han puesto a punto técnicas moleculares de detección que se basan en la identificación de alguna región de su genoma. Estas regiones se mantienen conservadas porque codifican proteínas que son esenciales en algún momento del ciclo biológico del virus. Una de estas técnicas es la PCR, y en concreto su variante a tiempo real puede detectar un número muy pequeño de virus, sólo unas decenas, en muestras infectadas. Este es el método de preferencia o “gold standard” para la detección sensible y específica de estos patógenos.
Sin embargo, debido a la diversidad genética que tiene una especie viral, es decir la multitud de cambios en el genoma que hay entre aislados de un mismo virus (mutaciones que lo hacen evolucionar) esta metodología no es perfecta y en ocasiones la región seleccionada para la detección por PCR no se encuentra conservada, lo que resulta en la aparición de falsos negativos.
La colaboración iniciada con el IVIA tenía por objetivo optimizar o desarrollar técnicas de RT-PCR a tiempo real para los virus de vid y así completar de forma eficaz la detección de los virus considerados en el Reglamento Técnico de Control y Certificación de Plantas de Vivero de Vid. En este contexto destaca el caso de la problemática en la detección del virus del enrollado 1 (GLRaV-1).
Las técnicas de RT-PCR convencionales y su variante en tiempo real, descritas hasta el momento, daban lugar a muchos falsos negativos, ya que no eran capaces de detectar diferentes aislados de origen geográfico diverso. El desarrollo de una nueva técnica de PCR a tiempo real para la detección de GLRaV-1 se realizó en las siguientes etapas:
1.- Análisis de secuencias genómicas de GLRaV-1 y diseño del método de PCR
Se analizaron todas las secuencias genómicas del virus del enrollado 1 disponibles en las bases de datos, así como secuencias genómicas obtenidas en el Laboratorio de Virología de Leñosas del IVIA mediante el análisis por secuenciación masiva de plantas de vid infectadas.
Este análisis permitió encontrar nuevas regiones genómicas conservadas en todos los aislados conocidos. Se diseñó un nuevo protocolo de PCR basado en la detección del virus en la zona del genoma responsable de producir las proteínas de la cápsida del virus, la envuelta proteica que protege su información genética.
2.- Validación de la técnica desarrollada para la detección de GLRaV-1.
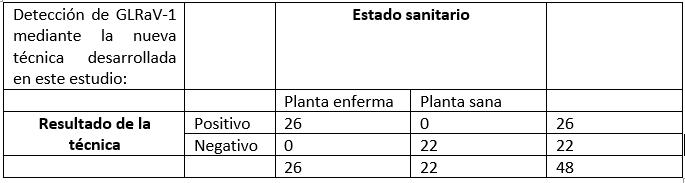
En una primera etapa se comprobó experimentalmente la capacidad de la técnica para detectar con éxito un total de 26 controles positivos procedentes de diferentes países (Suiza, Eslovaquia, Grecia, Túnez y España) infectadas con diferentes aislados virales de GLRaV-1. Como controles negativos se utilizaron 10 muestras de vid saneadas de la colección del IVIA y 12 muestras de vid infectadas con otros virus diferentes (incluyendo otros virus del enrollado GLRaV-2 y GLRaV-3, entrenudo corto infeccioso GLFV, vitivirus A, B y D, virus del jaspeado GFkV, virus del mosaico del arabis ArMV y los viroides HSVd y GYSVd-1) que no fueron detectados en ningún caso por la técnica.
En una segunda etapa se realizó la validación formal de la técnica, calculando los parámetros diagnósticos. Se estudiaron sensibilidad, especificidad, tasa de falsos positivos, tasa de falsos negativos, valor predictivo positivo y valor predictivo negativo de la técnica.
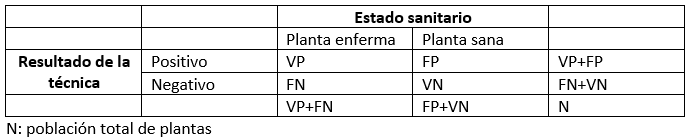
Para entender estos parámetros, se realiza una tabla de contingencia como la siguiente, en la que tendremos cuatro tipos de individuos: verdadero positivo (VP), planta infectada que la técnica diagnostica como positivo; falso positivo (FP), planta sana que la técnica diagnostica como enferma; falso negativo (FN), planta enferma no detectada por la técnica; y verdadero negativo (VN), planta sana que da un resultado negativo.
2.1.- Sensibilidad y especificidad diagnóstica
Estos dos indicadores de la capacidad operativa de una técnica, la sensibilidad y la especificidad, proporcionan información sobre la proporción de verdaderos positivos y verdaderos negativos identificados en la población de plantas enfermas o en la población de plantas sanas, respectivamente.
- El valor de sensibilidad equivale a VP/(VP+FN), es decir la proporción de plantas enfermas que han dado un resultado positivo.
- El valor de especificidad equivale a VN/(FP+VN), es decir, la proporción de plantas sanas que han dado un resultado negativo.
2.2.- Tasas de falsos positivos y falsos negativos
Estos dos indicadores muestran la proporción de falsos positivos y negativos en las poblaciones de plantas sanas y plantas enfermas respectivamente.
- La tasa de falsos positivos se puede calcular como FP / (FP + VN), que es la proporción de plantas sanas que han dado resultado positivo.
- La tasa de falsos negativos se calcula como FN / (VP + FN), que es la proporción de plantas enfermas que han dado un resultado negativo.
2.3.- Valores predictivos positivo y negativo
Estos dos parámetros proporcionan información sobre la probabilidad de que un resultado positivo o negativo de la técnica sea realmente positivo o negativo, respectivamente.
- El valor predictivo positivo equivale a VP/(VP+FP), es decir, la proporción de resultados positivos de la técnica que corresponden a verdaderos positivos.
- El valor predictivo negativo se calcula como VN/(FN+VN), es la proporción de resultados negativos de la técnica que corresponden a verdaderos negativos.
2.4.- Comparación entre la técnica disponible anteriormente y la desarrollada en este proyecto para la detección de GLRaV-1 a tiempo real
Para validar una nueva técnica se debe comparar con otra técnica establecida, reconocida y que se emplee como referente para la detección del virus concreto sobre el que se pone a punto el diagnóstico. En este caso la comparación se realizó con la PCR a tiempo real basada en la metodología a tiempo real de Osman y colaboradores publicada en 2007.
Detección de GLRaV-1 mediante la metodología de Osman y col. (2007)
Sensibilidad: 53%; Especificidad: 100%; Tasa de falsos negativos: 46%; Tasa de falsos positivos: 0%; Valor predictivo positivo: 100%; Valor predictivo negativo: 65%
Sensibilidad: 100%; Especificidad 100%; Tasa de falsos negativos: 0%; Tasa de falsos positivos: 0%; Valor predictivo positivo: 100%; Valor predictivo negativo: 100%.
En conclusión, la mejora en el diagnóstico de este virus ha sido excelente, y ha sido aplicada con éxito a muestras vegetales de prospecciones realizadas en campo, por lo que esta actualización será de enorme importancia para el control de este patógeno.
Agromillora dispone de un Laboratorio de Control de Calidad equipado para realizar RT-PCR de numerosos microorganismos, además de un plan de control sanitario que permite asegurar la calidad sanitaria de los diferentes productos que produce, por lo que el desarrollo de esta metodología será fácilmente aplicable para el análisis del estado sanitario de las plantas de vid a gran escala.
Referencia: Osman, F., Leutenegger, C., Golino, D., & Rowhani, A. (2007). Real-time RT-PCR (TaqMan) assays for the detection of Grapevine Leafroll associated viruses 1-5 and 9. Journal of Virological Methods, 141, 22-29.